解锁DNA-蛋白互作研究新技术,快来围攻表观遗传的新秀
染色质可及性
真核生物的核DNA并不是裸露的,而是有蛋白质即组蛋白与之相结合的。DNA一圈一圈地缠绕在组蛋白上,形成串珠式的结构。而这样的结构还能够进一步折叠、浓聚,并在其他架构蛋白的辅助下,形成染色体。DNA的复制,基因的转录,需要将DNA的高级结构解开。但是,这里的解开不需要将整个染色体全部打开,而只需要打开一部分,即需要表达基因的区域即可。而这一个过程,主要由染色体的表观修饰来实现的。这部分打开的染色质,就叫开放染色质(open chromatin)。而染色质一旦打开,就允许一些调控蛋白(比如转录因子和辅因子)跑过来与之相结合。而染色质的这种特性,就叫做染色质的可进入性或可及性(chromatin accessibility),如图1所示。
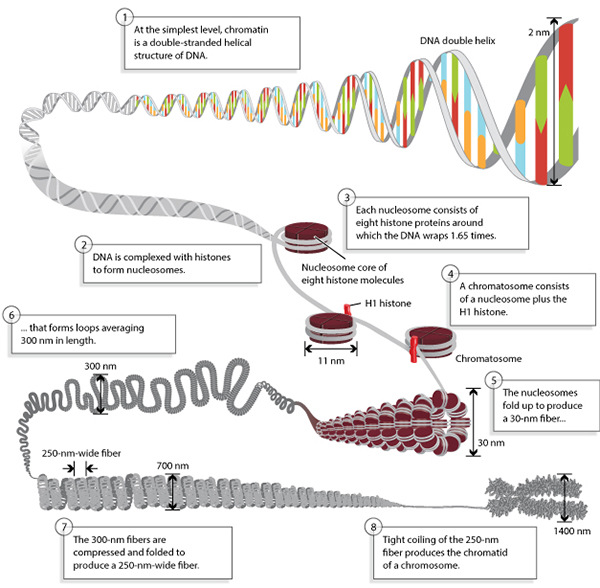
图1 DNA折叠与开放示意图
表观遗传学&研究方法
表观遗传学是一门研究基因表达调控的科学,它关注的是在不改变DNA序列的情况下,基因表达如何受到行为、环境和表型的影响,也就是说染色质的可及性对于基因表达的影响非常关键。表观遗传学的研究背景主要是对基因表达调控的深入理解,以及探索环境因素如何影响基因的活性和表达。
表观遗传学的研究方法包括但不限于以下几种:
1. ATAC-seq技术:这是一种用于分析染色质可及性的方法,可以揭示基因组中开放区域,这些区域通常是转录因子结合的位点。
2. 全基因组亚硫酸氢盐测序(WGBS):这种方法可以检测全基因组水平上的DNA甲基化状态,是研究表观遗传学中DNA甲基化的重要手段。
3. 简化代表性亚硫酸氢盐测序(RRBS):这是一种针对基因组中富含CpG岛区域的DNA甲基化分析方法,相对于WGBS,它成本较低。
4. 甲基化DNA免疫共沉淀测序(MeDIP-seq):通过使用抗甲基化DNA的抗体富集甲基化DNA片段,然后进行测序,以研究甲基化模式2。
5. 染色质免疫共沉淀测序(ChIP-seq):通过使用特定抗体富集与组蛋白修饰或转录因子结合的DNA片段,然后进行测序,用于研究蛋白质-DNA相互作用。
6. CUT&Tag技术:这是一种较新的技术,用于在单细胞水平上研究蛋白质-DNA相互作用,相较于ChIP-seq,它更为简便且适用于少量细胞样本2。
7. 3C-seq技术:这是一种用于研究染色质三维结构的技术,可以揭示基因组中不同区域的空间相互作用。
8. RNA测序(RNA-seq):通过测序RNA分子来研究基因表达水平,是研究非编码RNA和基因表达调控的重要方法。
这些方法的应用和普及改变了人们对多种表观遗传现象的传统认识,使研究人员能够更加全面地深入了解各种表观遗传标志在机体内的广泛分布,以及它们如何在外界因素的影响下发生相应的动态变化。其中ATAC和Cut&Tag由于其灵敏度高,操作方便等优点,成为研究表观遗传学的利器。下面我们就这两种方法来进行详细的介绍。
ATAC-seq技术详解
ATAC-seq
ATAC(Assay for Transposase-Accessible Chromatin using sequencing)是一种用于研究染色质可及性的高通量测序技术。它基于的原理是,染色质的开放区域更容易被转录因子和其他调控蛋白访问,这些区域通常与基因的活跃表达相关。该方法通过Tn5转座酶将序列接头插入到染色质开放区域,然后通过DNA测序数据分析来推断染色质各区域的基因活跃度情况。
ATAC-seq的关键步骤:
1. 样本准备:首先从细胞中提取染色质,这是DNA和组蛋白紧密结合形成的物质,它决定了基因的可访问性。
2. 染色质裂解:通过裂解细胞,释放出染色质,使其暴露出更多的表面区域。
3. 转座酶处理:使用一种特殊的转座酶Tn5,这种酶能够识别开放的染色质区域,并在这些区域切割DNA。
4. DNA片段化:转座酶在开放染色质区域切割DNA,产生小片段。
5. 测序接头插入:转座酶不仅切割DNA,还会在其切割位点插入测序所需的接头序列。
6. 高通量测序:对这些切割并带有接头的DNA片段进行高通量测序,以识别开放染色质区域。
7. 数据分析:测序数据通过生物信息学分析,确定开放染色质区域的位置,这些区域可能包含调控基因表达的关键元件。
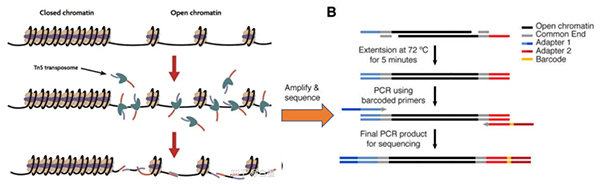
图2 ATAC实验原理示意图
CUT&Tag技术详解
CUT&Tag
CUT&Tag(Cleavage Under Targets and Tagmentation)是一种用于研究蛋白质-DNA相互作用的技术,该技术利用Tn5转座酶的特点,在切割染色质的同时直接插入接头(adaptor),极大地缩减了建库时间,且对样本量的要求更低,所需的测序深度也更低。
CUT&Tag的关键步骤:
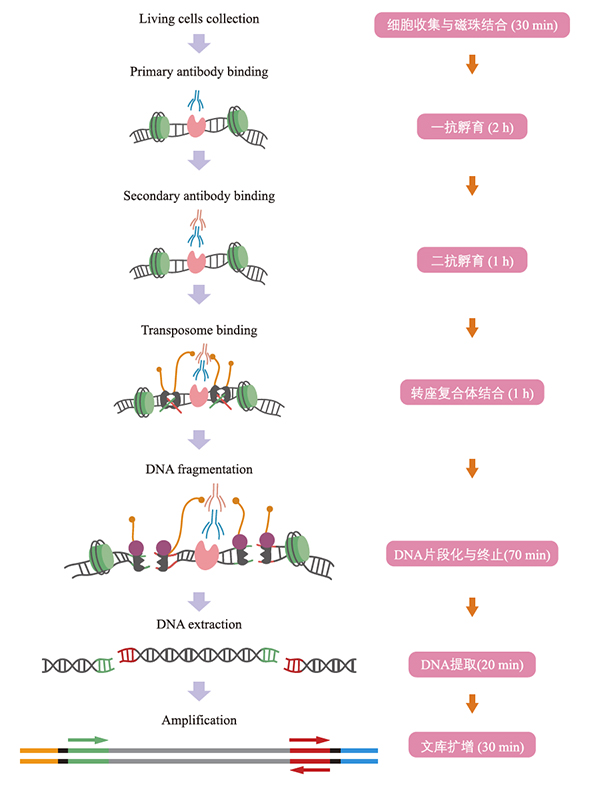
图3 Cut&Tag实验原理示意图
ATAC PK CUT&Tag
ATAC-seq和CUT&Tag是两种用于研究染色质状态和蛋白质-DNA相互作用的表观遗传学技术。它们在某些方面有相似之处,但也有明显的区别。以下是对这两种技术的详细比较:
ATAC-seq与CUT&Tag的联系:
ATAC-seq与CUT&Tag的区别:
|
|
ATAC-seq |
CUT&Tag |
研究重点 |
主要专注于分析染色质的开放性,即哪些区域对转录因子和其他调控蛋白是可及的。 |
更侧重于研究蛋白质-DNA相互作用,可以提供更详细的蛋白-染色质相互作用信息。 |
样本需求 |
通常需要较多的细胞样本,适用于大量细胞的分析。 |
特别适用于少量细胞样本,甚至可以在单细胞水平上进行分析。 |
实验流程 |
操作相对简单,主要涉及染色质的裂解和转座酶的切割。 |
需要使用特定抗体对蛋白进行标记,然后利用Tn5酶在抗体结合位点附近切割DNA。 |
技术灵敏度 |
适用于大规模的染色质开放性分析,但对于低丰度蛋白质的检测可能不够灵敏。 |
具有高灵敏度,能够检测低丰度蛋白质的染色质结合位点。 |
应用范围 |
广泛应用于研究启动子、增强子和其他调控元件的开放性变化。 |
适用于研究组蛋白修饰、转录因子结合位点和转录辅因子等,特别适合探究非典型DNA结构。 |
数据解读 |
数据解读相对直观,主要分析开放染色质区域。 |
数据解读可能更复杂,需要考虑蛋白质的特异性结合和相互作用。 |
结合使用这两种技术可以提供更全面的表观遗传学信息。ATAC-seq提供了染色质开放性的全局视图,而CUT&Tag则提供了特定蛋白质结合位点的详细信息。这种联合方法使研究人员能够同时研究染色质的可及性和特定蛋白质的结合模式,从而更深入地理解基因表达调控的复杂性。通过理解ATAC-seq和CUT&Tag的区别与联系,研究人员可以根据具体的研究目的和样本条件选择合适的方法,或者将两种技术联合使用,以获得更丰富的生物学信息。
经过以上介绍,相信大家已经对ATAC和Cut&Tag的研究方法以及它们之间的区别和联系有了一定的了解,目前这2种技术也被广泛的应用于表观遗传学的研究。全式金生物作为国产生物试剂原料供应商,深耕分子生物学领域多年,始终坚持自主创新,为客户提供优质的产品和服务。我们提供的ATAC、CUT&Tag建库试剂盒,建库效率高,测序数据质量好,助力表观遗传学研究。
产品推荐
ATAC
TransNGS® ATAC-Seq Library Prep Kit for Illumina®
产品特点
● 细胞数量少:所需细胞量少,50-50000细胞的起始量均可以获得高分辨率的测序图谱。
● 建库时间短:5000及以下细胞时,建库时间可缩短至2 h以内。
● 文库产量高:文库质量高,峰型好。
● 文库峰型好:文库峰型200-2000 bp,无明显接头峰。
● 无需分选:如对文库长度分布无特殊要求,可以不用分选。
CUT&Tag
TransNGS® CUT&Tag Library Prep Kit for Illumina®
产品特点
● 操作时间短:约5.5 小时即可完成文库构建。
● 测序数据质量高:文库产量高,建库峰型好。
● 数据准确可靠: clean data 占比高,mapping 率更稳定,peak 位点准确有效。
● 抗体兼容性好,亲和力高。
● 细胞投入范围广:适用于10-105 个哺乳动物细胞或经过处理的细胞核,以及无细胞壁结构的真核细胞(如植物细胞原生质体等)建库。
全式金生物提供的ATAC、CUT&Tag建库试剂盒,高效&精准&简便,助您轻松玩转表观遗传学研究,心动的小伙伴快来体验吧!
产品信息
|
产品名称 |
目录号 |
规格 |
|
KP171 |
12/96 rxns |
|
|
KP172 |
12/96 rxns |
参考文献:
DNA Packaging: Nucleosomes and Chromatin