我们知道,CUT&Tag和CUT&RUN技术都是Steven Henikoff开发的,2017年先发表了CUT&RUN,使用pA-MNase切割目标蛋白结合的DNA,得到的DNA需要进行末端修复加A加接头的过程。2019年发表的CUT&Tag中使用pA-Tn5转座子,切割后得到的DNA就已经添加了测序接头,大幅简化了操作步骤,因此更受到大家青睐。
那么为什么CUT&Tag没有完全取代CUT&RUN呢?除了建库流程的区别,其更深层次的原因是什么?接下来我们一起看看其在应用场景上的两个主要区别。
区别一:异染色质组蛋白修饰的适用性
先试想一下CUT&Tag与ATAC-seq的区别,两者都是使用Tn5酶切割染色质,核心区别在于是否使用了抗体富集。ATAC-seq中Tn5酶只切割开放的染色质,对处于关闭状态的异染色质区则不能进行切割,这也是做ATAC-seq的目的。那么如果要做异染色质组蛋白标记H3K9me3的分布位点,使用CUT&Tag合适吗?Tn5酶不是切割不了异染色质区吗?
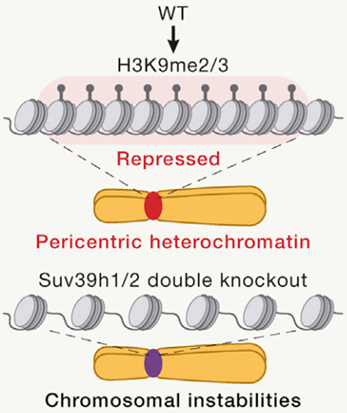
图1. 异染色质区组蛋白H3K9me2/3修饰[1]
从小编得到的信息来看,有一些老师使用CUT&Tag也能做出H3K9me3的位点,这其实是因为H3K9me3不是只分布在关闭区,有些将要关闭但还处在开放状态的染色质也可能会有H3K9me3的分布,再加上抗体介导的Tn5富集,关闭的区域也会有DNA能被转座到。
虽然如此,H3K9me3这种异染色质区域的组蛋白标记其实更适合用CUT&RUN,因为MNase酶的分子量更小,位阻更小,相比Tn5,能够更好的切割异染色质区的DNA,这也是为什么很多高分文章中H3K9me3的位点是用的CUT&RUN,这是其与CUT&Tag应用场景的第一个区别。

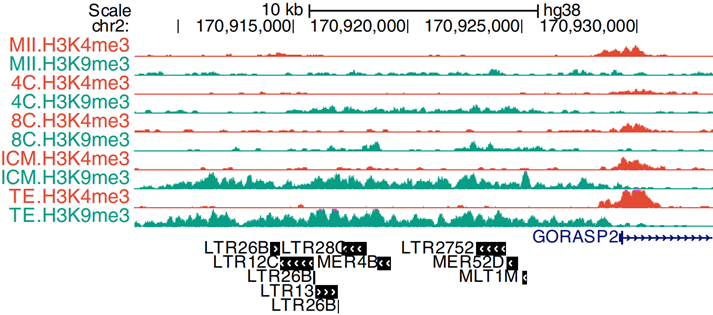
图2. CUT&RUN绘制人类早期胚胎发育过程中的H3K9me3位点[2]

图3. CUT&RUN检测斑马鱼胚胎发育过程中的H3K9me3位点[3]
区别二: 转录因子结合位点的分辨率
那么第二个区别是什么呢?答案是对于转录因子,CUT&RUN得到的结合位点具有更高的分辨率,尤其是先锋转录因子(pioneer transcription factors),一类能够直接与封闭染色质(缠绕在核小体上的DNA序列)结合并打开紧密的染色质结构的转录因子,进而增加染色质可及性并促进其它转录因子的结合,在早期启动基因转录过程中发挥重要作用。
Henikoff的原话:We prefer CUT&Tag for histone modifications as it does not require making sequencing libraries. However, we prefer CUT&RUN for transcription factors, as it provides base-pair resolution, which we have found very useful in our recent study of pioneer transcription factors (Meers et al. "Pioneer Factor-Nucleosome Binding Events during Differentiation Are Motif Encoded" Mol Cell. 2019 Jun 10.
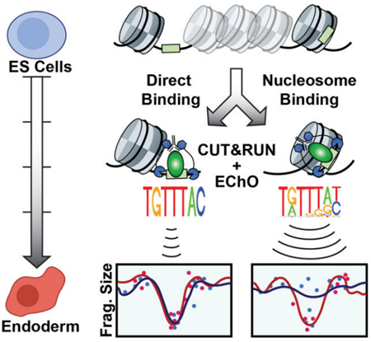
图4. CUT&RUN绘制先锋转录因子结合位点[4]
简单来说,CUT&RUN切割转录因子结合位点得到的片段范围更小,切割两端与结合位点中心更近,也就是碱基分辨率更高。当已经有目标基因时,可以通过设计引物进行qPCR定量分析目标蛋白的结合丰度在不同样本间的变化,这里以传统的ChIP-qPCR作为对比,文献中直接展示出了CUT&RUN-qPCR具有更好的富集和更高的分辨率[5]。

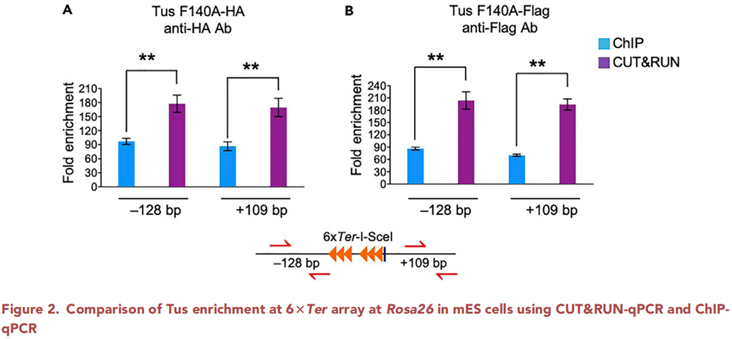
图5. 与ChIP-qPCR相比,CUT&RUN-qPCR具有更好的富集[5]
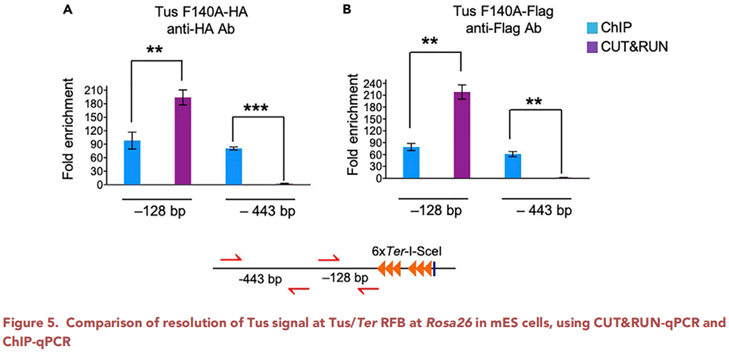
图6. 与ChIP-qPCR相比,CUT&RUN-qPCR具有更高的碱基分辨率[5]
总结一下,CUT&Tag的实验流程更简单,所需的细胞量可以比CUT&RUN更低,CUT&Tag适用于大多数非异染色质组蛋白的研究,也适用于多数转录因子的研究,其应用场景更广。而CUT&RUN更适用于异染色质区的组蛋白标记(H3K9me2/3)和先锋转录因子的研究。另外,由于对转录因子的分辨率更高,CUT&RUN-qPCR是传统ChIP-qPCR的理想替代者。
最后解释一个误区,有的老师可能会认为CUT&RUN对转录因子的成功率更高,实则不然。当一个常规的转录因子使用CUT&Tag建库失败时,排除操作的影响,那么再使用CUT&RUN大概率也不会成功,这种情况下,建议交联后再尝试或考虑更换抗体。实际上,针对转录因子通常会优先采用交联的方式,毕竟很多转录因子与DNA的结合并不是很牢固。
Vazyme 产品推荐
|
产品类别 |
产品名称 |
货号 |
规格 |
|
CUT&Tag |
Hyperactive Universal CUT&Tag Assay Kit for Illumina Pro |
12/48 rxns |
|
|
CUT&RUN |
Hyperactive pG-MNase CUT&RUN Assay Kit for PCR/qPCR |
24/48 rxns |
|
|
CUT&RUN |
Hyperactive pG-MNase CUT&RUN Assay Kit for Illumina |
24 rxns |
|
|
ATAC-seq |
Hyperactive ATAC-Seq Library Prep Kit for Illumina |
12/48 rxns |
参考文献:
[1] Haws, et al. 3D genome, on repeat: Higher-order folding principles of the heterochromatinized repetitive genome. Cell. 2022 Jul 21;185(15):2690-2707.
[2] Xu, et al. Stage-specific H3K9me3 occupancy ensures retrotransposon silencing in human pre-implantation embryos. Cell Stem Cell. 2022 Jul 7;29(7):1051-1066.e8.
[3] Akdogan-Ozdilek, et al. Identification of chromatin states during zebrafish gastrulation using CUT&RUN and CUT&Tag. Dev Dyn. 2022 Apr;251(4):729-742.
[4] Meers, et al. Pioneer Factor-Nucleosome Binding Events during Differentiation Are Motif Encoded. Mol Cell. 2019 Aug 8;75(3):562-575.e5.
[5] Panday, et al. A modified CUT&RUN-seq technique for qPCR analysis of chromatin-protein interactions. STAR Protoc. 2022 Jul 31;3(3):101529.